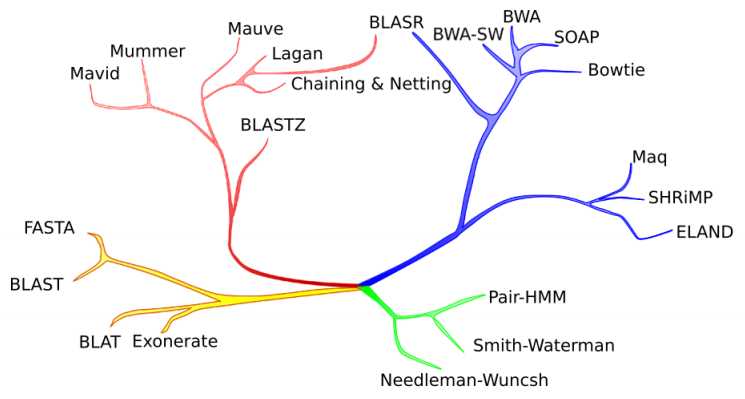
简单的局部比对:Blast、Blat等
formatdb -i db.seq [-options]
常用参数有以下几个:
-p:选择建库的类型,T表示蛋白库,F表示核酸库。缺省值为T
-o:判断是否分析序列名并建立序列名索引。T表示建立序列名索引,F表示不建立序列名索引。缺省值为F
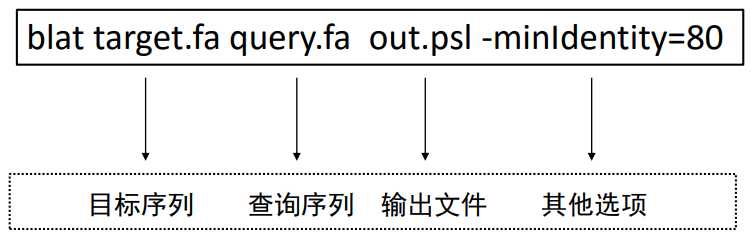
BLAST命令行:

输出结果:
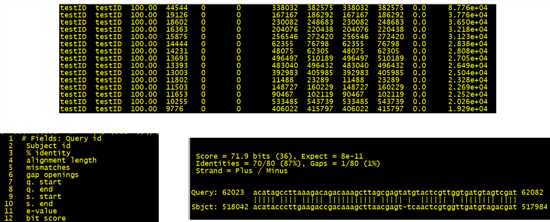
3是相似度、4是能比对上的区域
全局比对:MUSCLE、MATT等
muscle -in <inputfile> -out <outputfile>
原文:http://www.cnblogs.com/freescience/p/7476347.html