1 cutadapt -a AGATCGGAAGAGCACACGTCT -m 15 -q 20 --discard-untrimmed -o outname .fa
--discard-untrimmed 把reads中不含有adaper的reads去掉
-a 剪切reads 3‘端adapter(双端测序第一条read),加$表示adapter锚定在reads3‘端可找公司要
-g 剪切reads 5‘端adapter(双端测序第一条read),加$表示adapter锚定在reads 5‘端
-q 低质量碱基
-m readsd短于15时,丢弃该reads
我做的是植物。选择的miRNA长度为 15-30 bp
可自行写脚本进行统计
用R进行作图,ggplot
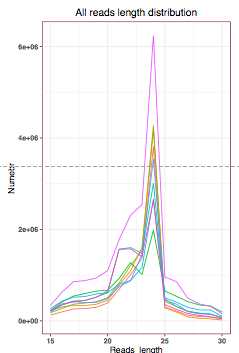
在24 中有一个峰值
原文:https://www.cnblogs.com/zhanmaomao/p/12150304.html