一.集成算法概述
集成学习(ensemble learning)是时下非常流行的机器学习算法,它本身不是一个单独的机器学习算法,而是通
过在数据上构建多个模型,集成所有模型的建模结果。基本上所有的机器学习领域都可以看到集成学习的身影,在
现实中集成学习也有相当大的作用,它可以用来做市场营销模拟的建模,统计客户来源,保留和流失,也可用来预
测疾病的风险和病患者的易感性。在现在的各种算法竞赛中,随机森林,梯度提升树(GBDT),Xgboost等集成
算法的身影也随处可见,可见其效果之好,应用之广。
集成算法的目标 :
集成算法会考虑多个评估器的建模结果,汇总之后得到一个综合的结果,以此来获取比单个模型更好的回归或
分类表现
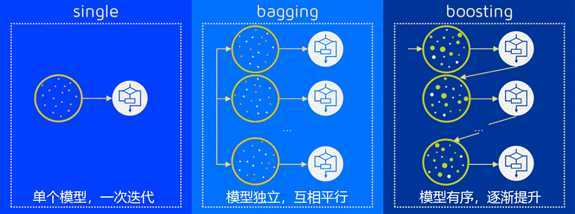
集成评估器
多个模型集成成为的模型叫做集成评估器(ensemble estimator),组成集成评估器的每个模型都叫做基评估器
(base estimator)。通常来说,有三类集成算法:装袋法(Bagging),提升法(Boosting)和stacking。
装袋法的核心思想是构建多个相互独立的评估器,然后对其预测进行平均或多数表决原则来决定集成评估器的结
果。装袋法的代表模型就是随机森林。
提升法中,基评估器是相关的,是按顺序一一构建的。其核心思想是结合弱评估器的力量一次次对难以评估的样本
进行预测,从而构成一个强评估器。提升法的代表模型有Adaboost和梯度提升树。
二.RandomForestClassifier---随机森林分类器
2.1重要参数
2.1.1控制基评估器的参数

2.1.2 n_estimators
这是森林中树木的数量,即基评估器的数量。这个参数对随机森林模型的精确性影响是单调的,n_estimators越
大,模型的效果往往越好。但是相应的,任何模型都有决策边界,n_estimators达到一定的程度之后,随机森林的
精确性往往不在上升或开始波动,并且,n_estimators越大,需要的计算量和内存也越大,训练的时间也会越来越
长。对于这个参数,我们是渴望在训练难度和模型效果之间取得平衡。
n_estimators的默认值在现有版本的sklearn中是10,但是在即将更新的0.22版本中,这个默认值会被修正为
100。这个修正显示出了使用者的调参倾向:要更大的n_estimators
创建一片随机森林
1.导入包
%matplotlib inline from sklearn.tree import DecisionTreeClassifier from sklearn.ensemble import RandomForestClassifier from sklearn.datasets import load_wine
2.导入红酒数据集
wine = load_wine()
wine.data
wine.target
3.sklearn建模的基本流程
from sklearn.model_selection import train_test_split Xtrain, Xtest, Ytrain, Ytest = train_test_split(wine.data,wine.target,test_size=0.3) clf = DecisionTreeClassifier(random_state=0) rfc = RandomForestClassifier(random_state=0) clf = clf.fit(Xtrain,Ytrain) rfc = rfc.fit(Xtrain,Ytrain) score_c = clf.score(Xtest,Ytest) score_r = rfc.score(Xtest,Ytest) print("Single Tree:{}".format(score_c) ,"Random Forest:{}".format(score_r) )
4. 画出随机森林和决策树在一组交叉验证下的效果对比
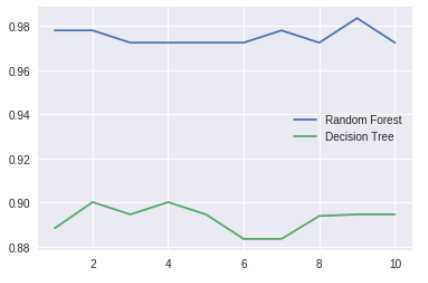
from sklearn.model_selection import cross_val_score import matplotlib.pyplot as plt rfc = RandomForestClassifier(n_estimators=25) rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10) clf = DecisionTreeClassifier() clf_s = cross_val_score(clf,wine.data,wine.target,cv=10) plt.plot(range(1,11),rfc_s,label = "RandomForest") plt.plot(range(1,11),clf_s,label = "Decision Tree") plt.legend() plt.show()
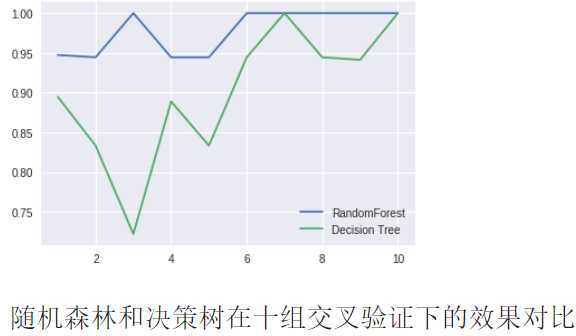
5. 画出随机森林和决策树在十组交叉验证下的效果对比
rfc_l = [] clf_l = [] for i in range(10): rfc = RandomForestClassifier(n_estimators=25) rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10).mean() rfc_l.append(rfc_s) clf = DecisionTreeClassifier() clf_s = cross_val_score(clf,wine.data,wine.target,cv=10).mean() clf_l.append(clf_s) plt.plot(range(1,11),rfc_l,label = "Random Forest") plt.plot(range(1,11),clf_l,label = "Decision Tree") plt.legend() plt.show() #是否有注意到,单个决策树的波动轨迹和随机森林一致? #再次验证了我们之前提到的,单个决策树的准确率越高,随机森林的准确率也会越高
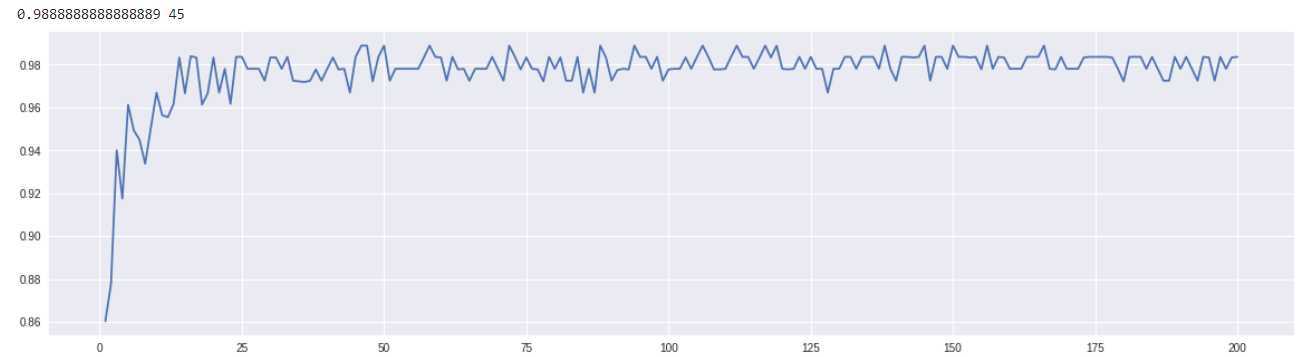
6. n_estimators的学习曲线
#####【TIME WARNING: 2mins 30 seconds】##### superpa = [] for i in range(200): rfc = RandomForestClassifier(n_estimators=i+1,n_jobs=-1) rfc_s = cross_val_score(rfc,wine.data,wine.target,cv=10).mean() superpa.append(rfc_s) print(max(superpa),superpa.index(max(superpa))) plt.figure(figsize=[20,5]) plt.plot(range(1,201),superpa) plt.show()
2.1.3 random_state
随机森林中其实也有random_state,用法和分类树中相似,只不过在分类树中,一个random_state只控制生成一
棵树,而随机森林中的random_state控制的是生成森林的模式,而非让一个森林中只有一棵树。
estimators属性:查看森林中树的状况
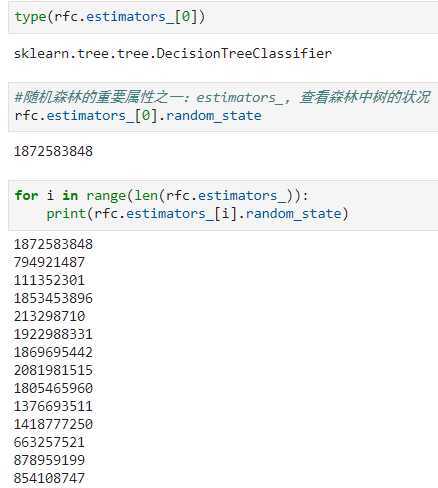
当random_state固定时,随机森林中生成是一组固定的树,但每棵树依然是不一致的,这是
用”随机挑选特征进行分枝“的方法得到的随机性。并且我们可以证明,当这种随机性越大的时候,袋装法的效果一
般会越来越好。用袋装法集成时,基分类器应当是相互独立的,是不相同的。
但这种做法的局限性是很强的,当我们需要成千上万棵树的时候,数据不一定能够提供成千上万的特征来让我们构
筑尽量多尽量不同的树。因此,除了random_state。我们还需要其他的随机性。
2.1.4 bootstrap & oob_score
要让基分类器尽量都不一样,一种很容易理解的方法是使用不同的训练集来进行训练,而袋装法正是通过有放回的
随机抽样技术来形成不同的训练数据,bootstrap就是用来控制抽样技术的参数。
在一个含有n个样本的原始训练集中,我们进行随机采样,每次采样一个样本,并在抽取下一个样本之前将该样本
放回原始训练集,也就是说下次采样时这个样本依然可能被采集到,这样采集n次,最终得到一个和原始训练集一
样大的,n个样本组成的自助集。由于是随机采样,这样每次的自助集和原始数据集不同,和其他的采样集也是不
同的。这样我们就可以自由创造取之不尽用之不竭,并且互不相同的自助集,用这些自助集来训练我们的基分类
器,我们的基分类器自然也就各不相同了。
bootstrap参数默认True,代表采用这种有放回的随机抽样技术。通常,这个参数不会被我们设置为False。
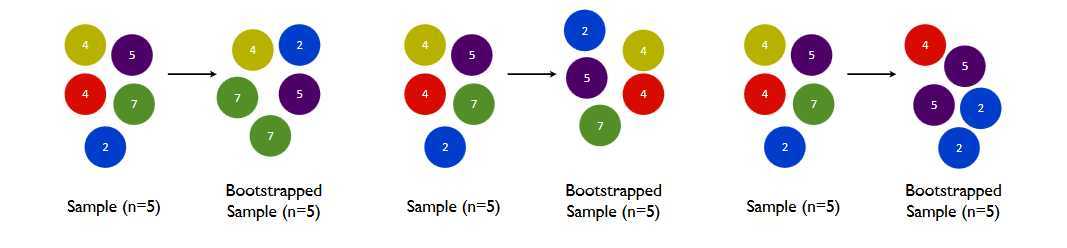
然而有放回抽样也会有自己的问题。由于是有放回,一些样本可能在同一个自助集中出现多次,而其他一些却可能
被忽略,一般来说,自助集大约平均会包含63%的原始数据因此,会有约37%的训练数据被浪费掉,没有参与建模,
这些数据被称为袋外数据(out of bag data,简写为oob)。除了我们最开始就划分好的测试集之外,这些数据也可
以被用来作为集成算法的测试集。也就是说,在使用随机森林时,我们可以不划分测试集和训练集,只需要用袋外
数据来测试我们的模型即可。当然,这也不是绝对的,当n和n_estimators都不够大的时候,很可能就没有数据掉
落在袋外,自然也就无法使用oob数据来测试模型了。
如果希望用袋外数据来测试,则需要在实例化时就将oob_score这个参数调整为True,训练完毕之后,我们可以用
随机森林的另一个重要属性:oob_score_来查看我们的在袋外数据上测试的结果:
#无需划分训练集和测试集 rfc = RandomForestClassifier(n_estimators=25,oob_score=True) rfc = rfc.fit(wine.data,wine.target) #重要属性oob_score_ rfc.oob_score_
2.2 重要属性和接口
决策树的参数,并通过n_estimators,
random_state,boostrap和oob_score这四个参数帮助大家了解了袋装法的基本流程和重要概念。同时,我们还
介绍了.estimators_ 和 .oob_score_ 这两个重要属性。除了这两个属性之外,作为树模型的集成算法,随机森林
自然也有.feature_importances_这个属性。
随机森林的接口与决策树完全一致,因此依然有四个常用接口:apply, fit, predict和score。除此之外,还需要注
意随机森林的predict_proba接口,这个接口返回每个测试样本对应的被分到每一类标签的概率,标签有几个分类
就返回几个概率。如果是二分类问题,则predict_proba返回的数值大于0.5的,被分为1,小于0.5的,被分为0。
传统的随机森林是利用袋装法中的规则,平均或少数服从多数来决定集成的结果,而sklearn中的随机森林是平均
每个样本对应的predict_proba返回的概率,得到一个平均概率,从而决定测试样本的分类
#大家可以分别取尝试一下这些属性和接口 rfc = RandomForestClassifier(n_estimators=25) rfc = rfc.fit(Xtrain, Ytrain) rfc.score(Xtest,Ytest) rfc.feature_importances_ rfc.apply(Xtest) rfc.predict(Xtest) rfc.predict_proba(Xtest)
三.RandomForestRegressor
所有的参数,属性与接口,全部和随机森林分类器一致。仅有的不同就是回归树与分类树的不同,不纯度的指标,
参数Criterion不一致。
3.1 重要参数,属性与接口
criterion
回归树衡量分枝质量的指标,支持的标准有三种:
1)输入"mse"使用均方误差mean squared error(MSE),父节点和叶子节点之间的均方误差的差额将被用来作为
特征选择的标准,这种方法通过使用叶子节点的均值来最小化L2损失
2)输入“friedman_mse”使用费尔德曼均方误差,这种指标使用弗里德曼针对潜在分枝中的问题改进后的均方误差
3)输入"mae"使用绝对平均误差MAE(mean absolute error)。
在回归树中,MSE不只是我们的分枝质量衡量指标,也是我们最常用的衡
量回归树回归质量的指标,当我们在使用交叉验证,或者其他方式获取回归树的结果时,我们往往选择均方误差作
为我们的评估(在分类树中这个指标是score代表的预测准确率)。在回归中,我们追求的是,MSE越小越好。
然而,回归树的接口score返回的是R平方,并不是MSE。
R平方可以为正为负(如果模型的残差平方和远远大于
模型的总平方和,模型非常糟糕,R平方就会为负),而均方误差永远为正。
值得一提的是,虽然均方误差永远为正,但是sklearn当中使用均方误差作为评判标准时,却是计算”负均方误
差“(neg_mean_squared_error)。这是因为sklearn在计算模型评估指标的时候,会考虑指标本身的性质,均
方误差本身是一种误差,所以被sklearn划分为模型的一种损失(loss),
因此在sklearn当中,都以负数表示。真正的
均方误差MSE的数值,其实就是neg_mean_squared_error去掉负号的数字。
重要属性和接口
最重要的属性和接口,都与随机森林的分类器相一致,还是apply, fit, predict和score最为核心。值得一提的是,随
机森林回归并没有predict_proba这个接口,因为对于回归来说,并不存在一个样本要被分到某个类别的概率问
题,因此没有predict_proba这个接口。
随机森林回归用法
和决策树完全一致,除了多了参数n_estimators
from sklearn.datasets import load_boston from sklearn.model_selection import cross_val_score from sklearn.ensemble import RandomForestRegressor boston = load_boston() regressor = RandomForestRegressor(n_estimators=100,random_state=0) cross_val_score(regressor, boston.data, boston.target, cv=10 ,scoring = "neg_mean_squared_error") sorted(sklearn.metrics.SCORERS.keys())
返回十次交叉验证的结果,注意在这里,如果不填写scoring = "neg_mean_squared_error",交叉验证默认的模型
衡量指标是R平方,因此交叉验证的结果可能有正也可能有负。而如果写上scoring,则衡量标准是负MSE,交叉验
证的结果只可能为负
原文:https://www.cnblogs.com/zxc7/p/12830785.html