朴素贝叶斯算法依据概率论中贝叶斯定理建立模型,前提假设各个特征之间相互独立(这也是正式“朴素”的含义),这个假设非常极端,因为实际场景中多个特征一般存在相关性,特征相对独立的假设使得算法变得简单,因此在特征值有强相关性的场景中容易出现分类不准的问题。
其数学原理很容易理解:如果你看到一个人总是做好事,则会推断那个人多半会是一个好人。这就是说,当你不能准确判断时候,可以依靠事物特定本质相关的事件出现的多少(概率)作为判断依据,贝叶斯定理:
![]()
该公式表示在B发生的条件下A发生的条件概率,等于A事件发生条件下B事件发生的条件概率乘以A事件的概率,再除以B事件发生的概率。公式中,P(A)叫做先验概率,P(A/B)叫做后验概率。
举个栗子:一个非常炎热的夏天晚上,走在校园里面,伸手不见五指.......lol,这个时候迎面走来一个人,太远看不清楚ta的性别,但我们知道ta的特征是“短裤+短发”,而且事先有一些学生的调查样本,需要你根据某些特性大致判断Ta的性别,请问你应该怎么分类?

这样分析,我们首先计算求得P(boy|短裤短发)和P(girl|短裤短发)然后比较两者大小,作为依据判定性别,也就是我们根据以往数据中穿着短裤短发的人中boy和girl的条件概率作为依据,来判断当我们看见“短裤短发”人的性别,在这个例子中我们很明显把ta判定是个boy,核心思想就是这么简单:

由于特征空间较为稀疏,因此,常常会出现概率为0的情况,在这种情况下,需要对其进行一些修正。常用的修正方法是拉普拉斯修正法,就是使得计算条件概率时候分子+1,很容易理解;
该数据集包含了8124个样本和22个变量(如蘑菇的颜色、形状、光滑度等),是机器学习分类算法算法不可多得的一个优质数据集。
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import numpy as np
# 修改baseUrl的路径即可完成数据读取修改
baseUrl="C:\\Users\\71781\\Desktop\\2020\\ML-20200422\\bayes\\"
mushrooms=pd.read_csv(baseUrl+"mushrooms.csv")
mushrooms.columns=[‘class‘,‘cap-shape‘,‘cap-surface‘,‘cap-color‘,‘ruises‘,‘odor‘,‘gill-attachment‘,‘gill-spacing‘,‘gill-size‘,‘gill-color‘,‘stalk-shape‘,‘stalk-root‘,‘stalk-surface-above-ring‘,‘stalk-surface-below-ring‘,‘stalk-color-above-ring‘,‘stalk-color-below-ring‘,‘veil-type‘,‘veil-color‘,‘ring-number‘,‘ring-type‘,‘spore-print-color‘,‘population‘,‘habitat‘]
mushrooms.shape
pd.set_option("display.max_columns",100) #让所有列都能加载出来
mushrooms.head()
# 可以发现,所有特征都是离散的,都属于分类型
# class标识有毒无毒
np.unique(mushrooms[‘cap-shape‘])
fig,(ax1,ax2)=plt.subplots(1,2,figsize=(15,5))
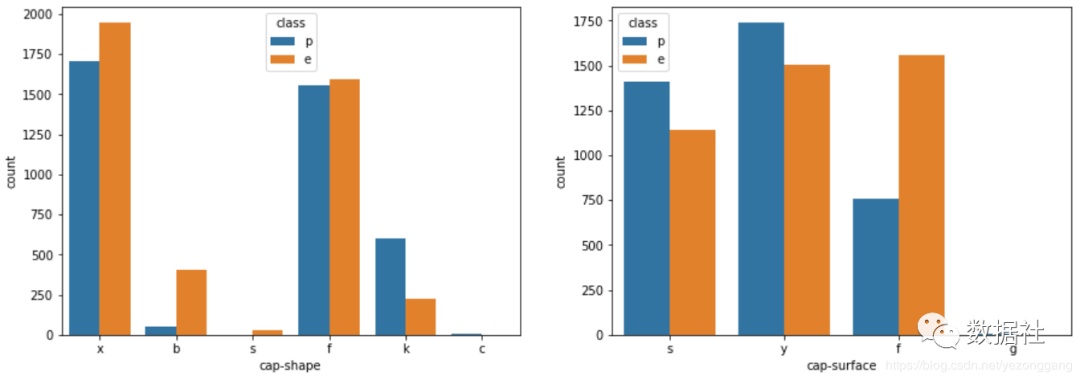
# 探究 形状和颜色对于是否有毒的贡献度,发现形状为b的无毒蘑菇比例大
sns.countplot(x=‘cap-shape‘,data=mushrooms,hue=‘class‘,ax=ax1)
sns.countplot(x=‘cap-surface‘,data=mushrooms,hue=‘class‘,ax=ax2)
sns.countplot(x=‘cap-color‘,hue=‘class‘,data=mushrooms)

# 把有毒无毒换成0/1类型,1标识无毒 mushrooms[‘class‘].replace(‘e‘,1,inplace=True) mushrooms[‘class‘].replace(‘p‘,0,inplace=True) # 计算每个颜色无毒的概率 perc = mushrooms[["cap-color", "class"]].groupby([‘cap-color‘],as_index=False).mean() perc sns.barplot(x=‘cap-color‘,y=‘class‘,data=perc)

# 使用sklearn进行预处理
from sklearn.preprocessing import LabelEncoder
labelencoder=LabelEncoder()
for col in mushrooms.columns:
mushrooms[col] = labelencoder.fit_transform(mushrooms[col])
mushrooms.head()
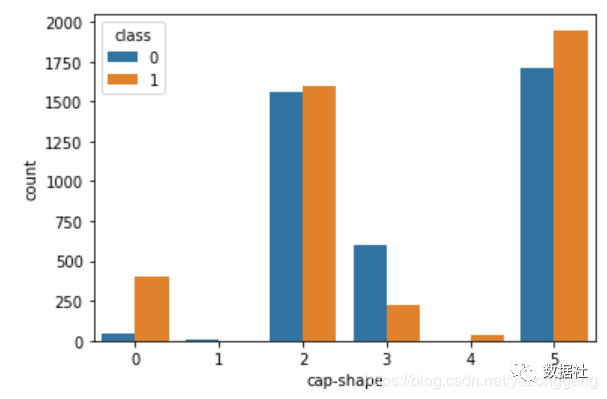
sns.countplot(x=‘cap-shape‘,data=mushrooms,hue=‘class‘,)
X=mushrooms.drop(‘class‘,axis=1) #Predictors
y=mushrooms[‘class‘] #Response
#X.head()
#这里采用用哑变量编码,为的是后面能更好的计算特征的各属性的重要性,并且避免数值变量分类时偏向于数值大的属性
X=pd.get_dummies(X,columns=X.columns,drop_first=True)
X.head()
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=1234)
# 贝叶斯
from sklearn.naive_bayes import GaussianNB
from sklearn import metrics
model2 = GaussianNB()
model2.fit(X_train, y_train)
prediction2 = model2.predict(X_test)
print(‘The accuracy of the Decision Tree is: {0}‘.format(metrics.accuracy_score(prediction2,y_test)))
原文:https://www.cnblogs.com/qinchaofeng/p/13729459.html